
SageMaker Pipelinesでアルゴリズムのバージョン変更時に、Processing Jobのエラーが発生原因の確認点を挙げてみた
データ事業本部の鈴木です。
SageMaker Pipelinesのチュートリアルはバージョンが古めのXGBoostモデルを使っていたため、アルゴリズム向けのコンテナのバージョンを上げてみましたが、変更とは関係のない後続のステップでエラーが発生したためデバッグしていました。
原因としてはフレームワークのバージョンを上げると訓練済みのモデルの読み込み方法が変わっていたなどの単純なものでしたが、実際使用する上で起こり得そうなことだったので、確認した内容をご共有します。
今回はモデルの読み込みとバッチ推論でエラーが発生しましたが、確認したポイントとしては
- Pipelineの実行履歴のメッセージとCloudWatch Logsに出力されるエラーの確認
- S3バケットにあるモデルアーティファクトの取得とエラーの再現
- アルゴリズムのバージョン差に起因する事象の調査
です。
発生した事象
XGBoostのアルゴリズムのバージョンを1.0-1から1.7-1に変更しました。
具体的には
image_uri = sagemaker.image_uris.retrieve(
framework="xgboost",
region=region,
version="1.0-1",
py_version="py3",
instance_type="ml.m5.xlarge",
)
を以下に修正しました。
image_uri = sagemaker.image_uris.retrieve(
framework="xgboost",
region=region,
version="1.7-1",
py_version="py3",
instance_type="ml.m5.xlarge",
)
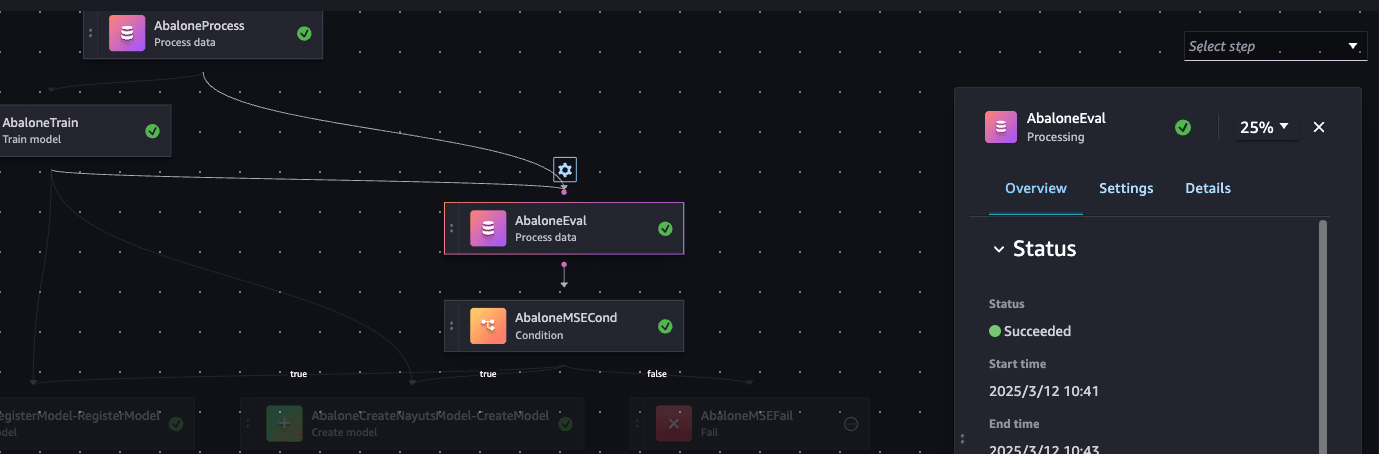
するとPipeline実行時に、以下のようにテスト用データセットでのメトリクス評価のステップで失敗しました。
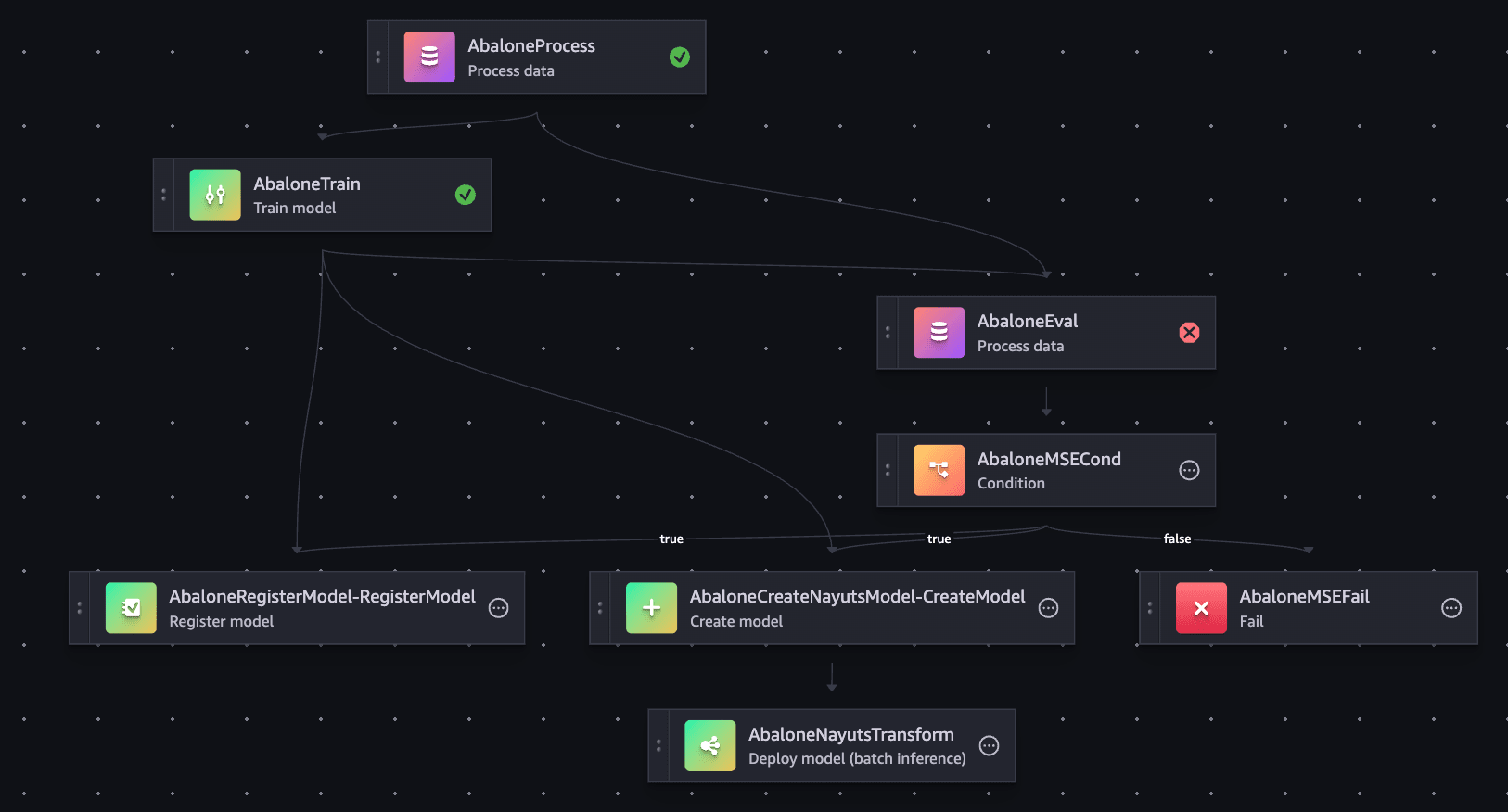
なお、コンテナのバージョンは以下を参照しています。
確認した箇所
1. メッセージとCloudWatch Logsのログ
まずは失敗が発生したステップで出力されているメッセージを確認しました。
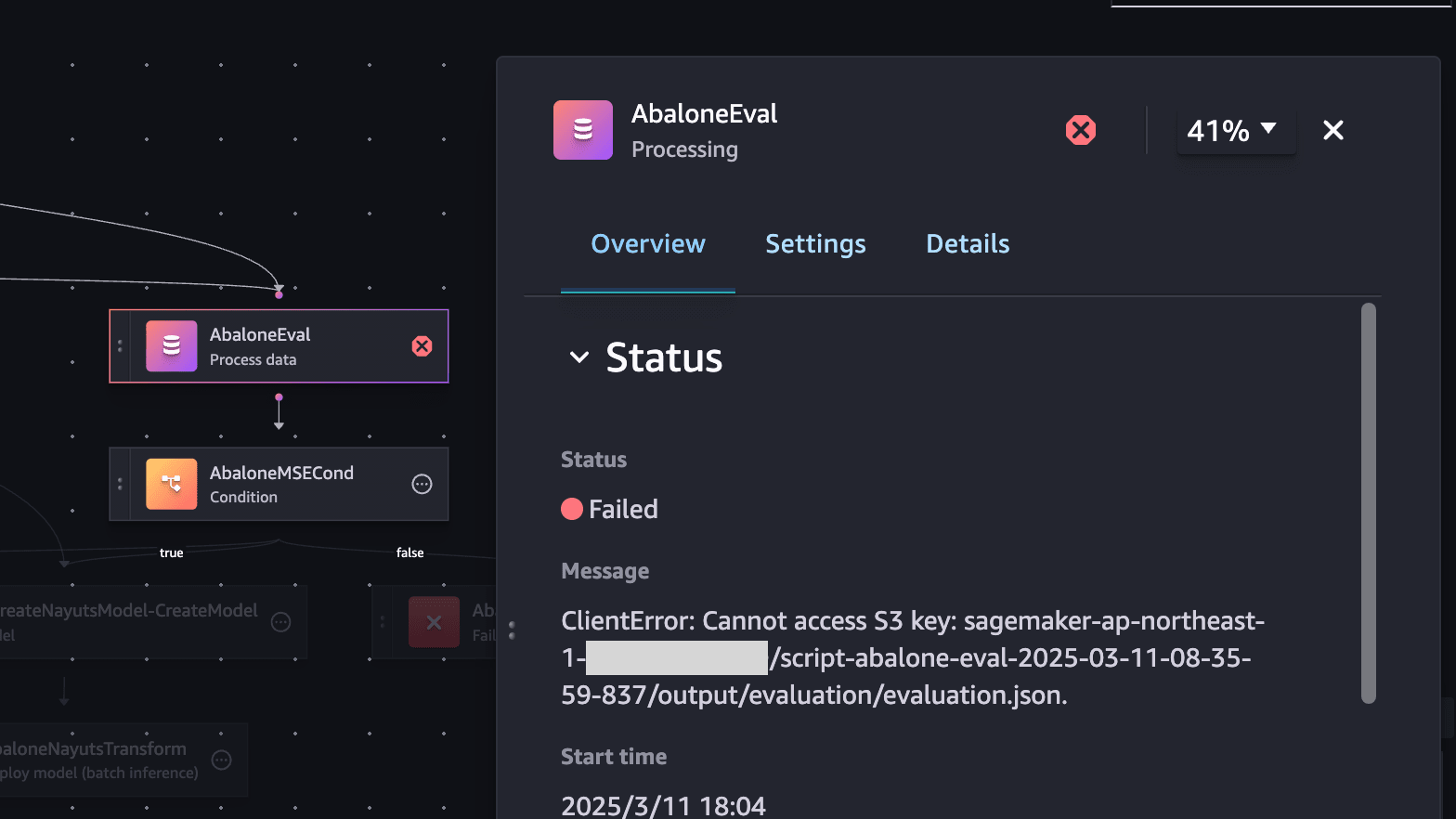
ここではS3バケットにJSONファイルがないというエラーが出ていました。S3バケットには確かに該当のオブジェクトはないのですが、あまり関係がなさそうだったのでもう少しほかの出力を確認しました。
DetailsにはCloudWatch Logsへのログが出ているため、確認しました。pickle.load(open("xgboost-model", "rb"))でエラーが出ていることが分かり、なにかモデルの読み込みで失敗していることが想像されました。
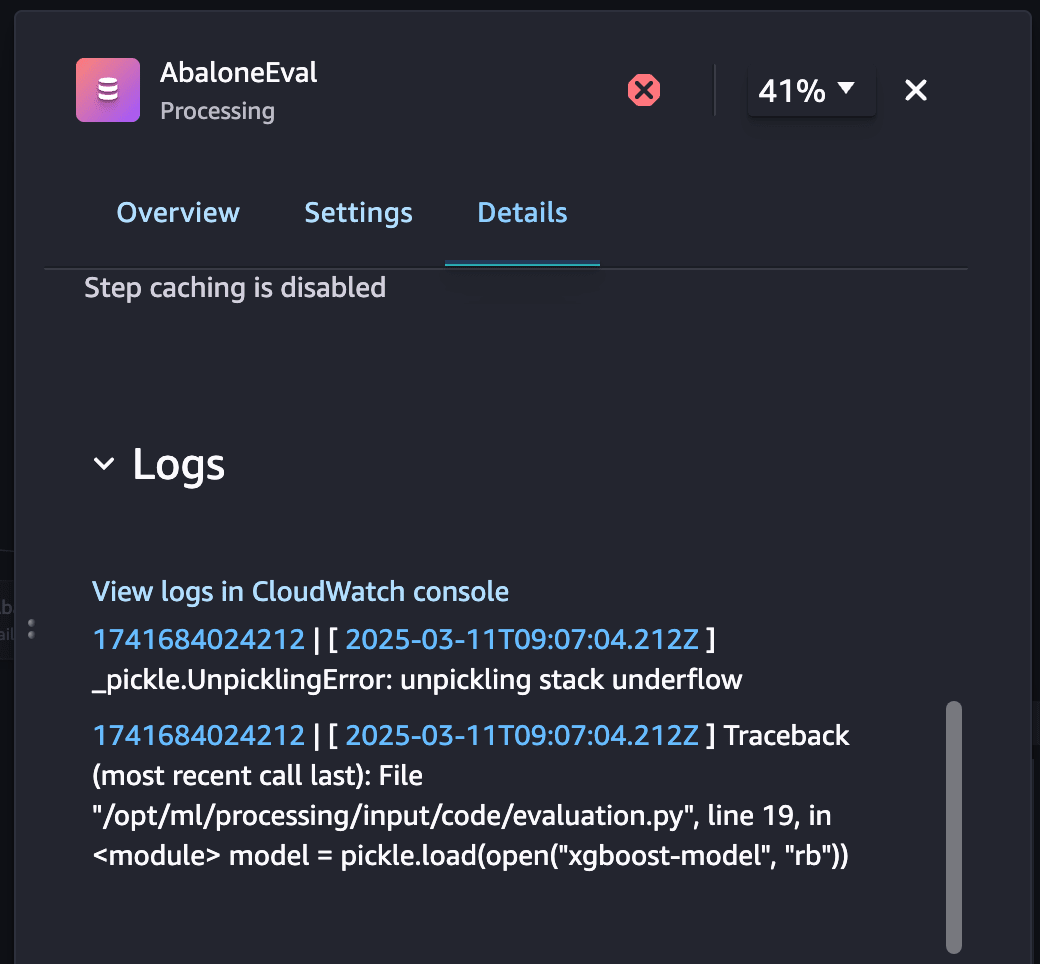
この読み込みの実装はProcessing Jobで実行するevaluation.pyの以下の実装にあたります。
model_path = f"/opt/ml/processing/model/model.tar.gz"
with tarfile.open(model_path) as tar:
tar.extractall(path=".")
model = pickle.load(open("xgboost-model", "rb"))
2. モデルアーティファクトの取得
モデル訓練のステップでは、S3バケットのどこにモデルアーティファクトが出力されているか記載されているため、オブジェクトを取得しました。
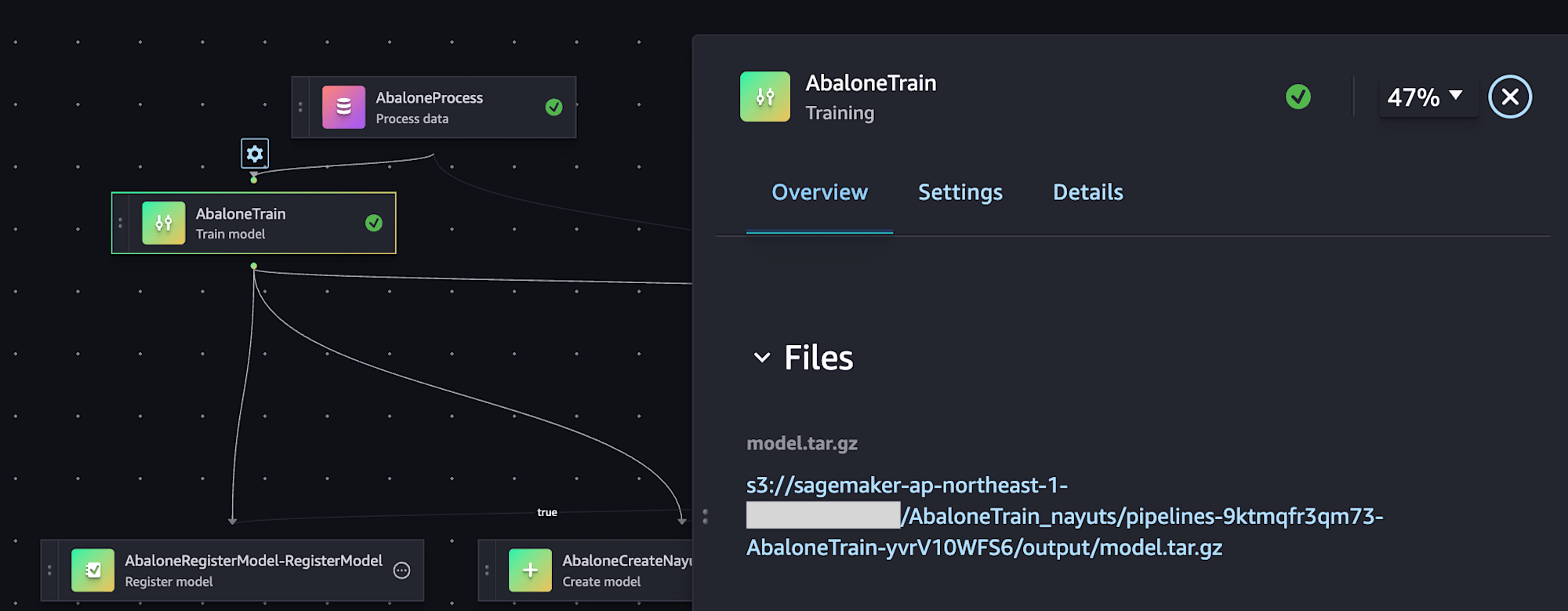
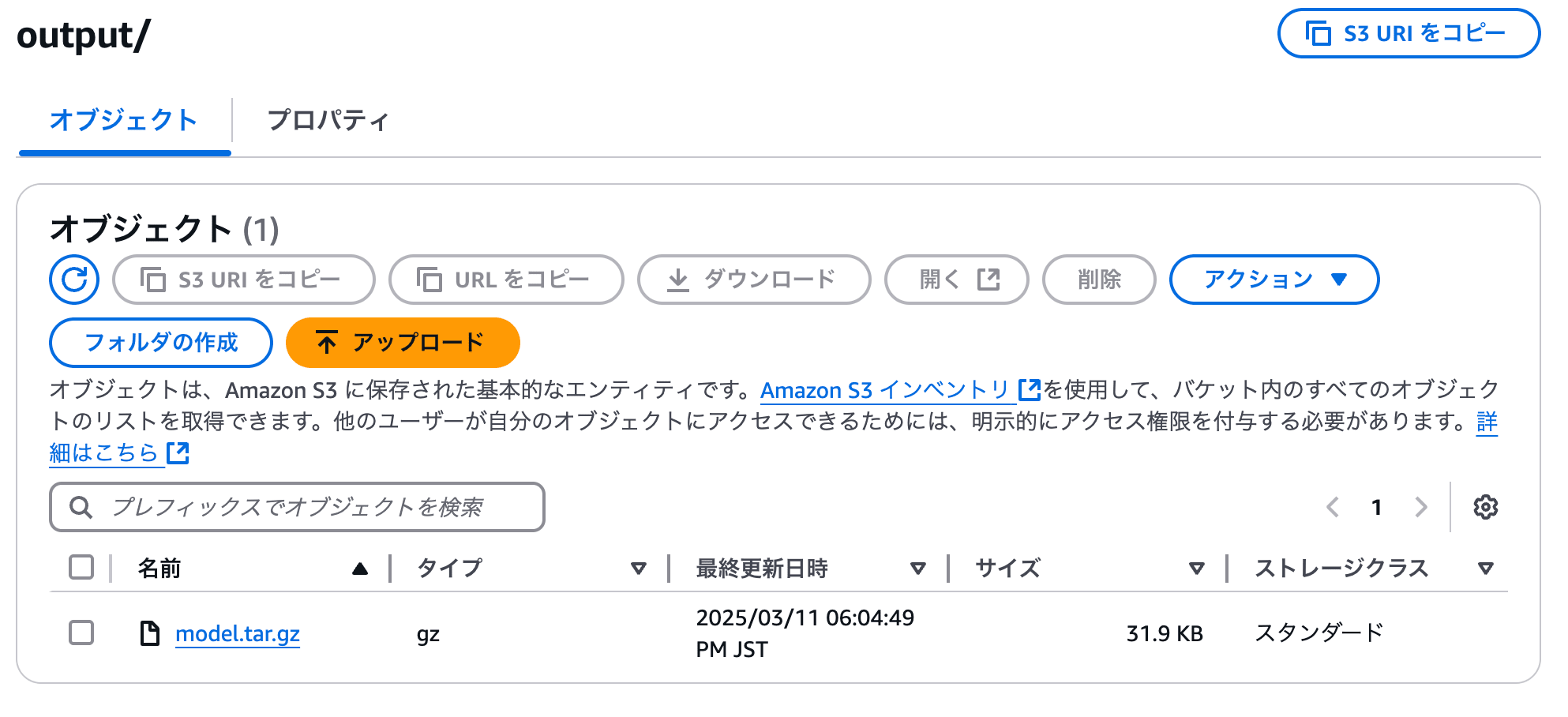
SageMaker StudioのJupyterLabを開き、モデルアーティファクトをコピーしてProcessing Jobで実行しているモデルの読み込みをすると、確かに同じエラーが発生することを確認しました。
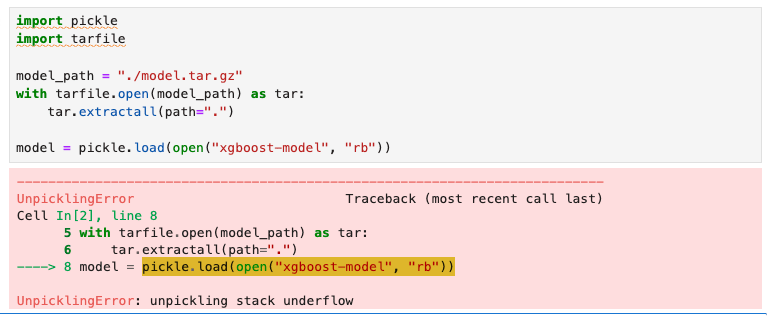
このエラーで検索すると、stack overflowで同様の事象の質問がありました。1.3以降のバージョンではモデルの読み込み方法が変わったことが原因でした。
3. Processing Jobの再実行
以下のようにモデルの読み込みを変更し、再度実行して、失敗したステップが成功することを確認しました。
model_path = f"/opt/ml/processing/model/model.tar.gz"
with tarfile.open(model_path) as tar:
tar.extractall(path=".")
model = xgboost.Booster()
model.load_model('xgboost-model')
補足
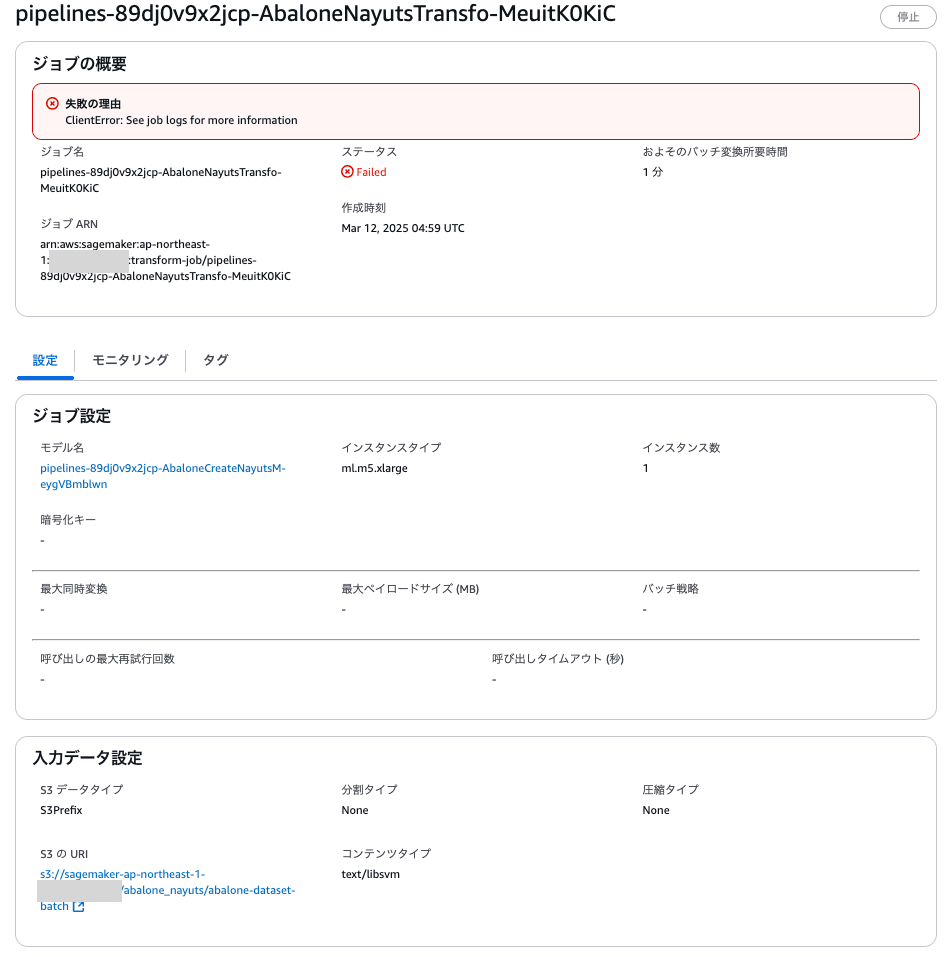
なお、このステップを通過しても、最後のバッチ変換ジョブでNumber of columns does not match number of features in booster.のエラーとなりました。
このステップではabalone-dataset-batchというlibsvmファイルを入力にバッチ推論を実行します。
原因としては、恐らく新しいバージョンのXGBoostのlibsvmファイルの読み込み方法が古いバージョンで行なっていたものと変わっていることが考えられました。バッチ変換ジョブで実行時にコンテンツタイプをtext/libsvmとしても同じエラーが発生しました。
以下はSageMakerのコンソールから確認した例になります。
ここは実装を変えて、バッチ変換ジョブの入力となるlibsvmファイルをCSVに変換するProcessing Jobを前に配置するようにするのがよさそうでした。
最後に
SageMaker Pipelines内のProcessing Jobのデバッグ例でした。同様の事象が起きた際に参考になりましたら幸いです。







